Holgonaut hat geschrieben:Hmm, weiß nicht, was an meinem Argument "zu einfach" war.
Sorry; das bezog sich lediglich auf dein knappes Kommentar zum Thema Informatonskriterien, denn auch bei konventionellen Fit indizes wird die Modell-Sparsamkeit in Form der Freiheitsgrade mit berücksichtigt. In Gegensatz dazu bewerten BIC, AIC etc. Modelle in Bezug auf ihren prädiktiven Wert.
Holgonaut hat geschrieben:Jetzt kommt es darauf an, was du mit Modellannahmen meinst. Es gibt eine Menge. Für mich zentral ist die Annahme der korrekten kausalen Spezifikation. Für Statistiker, die einen großen Teil der Literatur in SEM beitragen, ist das oft wenig Interesse, da sie sich lediglich eben mit statistischen Dingen wie Robustheit etc. beschäftigen. Für mich als inhaltlichen Forscher nur soweit von Belang, wie meine theoretische Perspektive bedroht ist.
Ok, sehen wir davon ab, dass die neure Entwicklung von Strukturgleichungsmodelle nicht in erster Linie durch Statistiker, sondern durch praktisch orientierte Methodologen der Sozio- und Psychologie vorangetrieben wurde (Curran, Bentler, McCallum, Bollen, McArdle ua.). Statistiker freuen sich, eher wie du, Holger, über Datenpassung. O.a. Gruppe geht es hingegen aber um die Spezifikation korrekter Kausalmodelle (die wiederrum bestimmte strukturelle Eigenschaften von Daten implizieren -> Modellannahmen). Du schreibst, dass es dir eigentlich auch darum ginge, aber deine Argumentation lässt eher einen anderen Schluss zu.. Wie du schreibst, sind Missspezifikationen nur dann für dich von Belang, wenn sie deine theoretische Perspektive bedrohen würden und genau da liegt der Hase im Pfeffer, denn überkomplexe Modelle tun genau das.
Gehen wir mal davon aus, dass du zwei fittende und sehr komplexe Modelle (mit identischen Freiheitsgraden) konstruierst. Eines ist faktisch (in Bezug auf die Abbildung der „wahren“, datengenerierenden Kausalbeziehungen) falsch spezifiziert, fittet aber sehr gut. Das andere „fittet mit“ Abstand schlechter, wäre aber faktisch korrekt. Wenn du nun diese hochkomplexen Modelle an einer anderen Stichprobe (siehe weiter unten, um Verwechselungen vorzubeugen) validieren möchtest, wäre die Chance relativ groß, dass du bei deinem favorisierten Model einen Missfit bekommst, was einzig und allein an (für den datengenerierenden Prozess) inadäquaten Parametern liegt, die nur deiner ursprünglichen Stichprobe den Fit verbessern. Jetzt gehen wir einen Schritt zurück und denken uns, dass du das 2., richtige, aber in deiner Stichprobe schlecht fittende Modell, gar nicht erst berichtest, weil du es verworfen hast und du es davon abgesehen nicht sinnvoll interpretieren konntest. Stattdessen berichtest du ein weniger komplexes Model als „preliminary“, dass nested in beiden kontrastierten Modellen war. Bei der Replikation wird sich nun zeigen, dass dieses preliminary modell deutlicher weniger fitness verliert, als dein Favorisiertes. Vielleicht fallen auch beide unter die Signifikanzschwelle, mit dem verworfenen wärs dir aber nicht so gegangen. Ich denke, es sollte klar werden, dass die Kausalstruktur dieses „sparsameren“ Modells dichter am Datengenerierenden Prozess ist, als dein missspezifiziertes, aber vermeintlich überlegenes Modell.
Holgonaut hat geschrieben:Und zur Überprüfung der kausalen Spezifikation ist die Replikation (auch wenn das häufig so gesehen wird), kein Königsweg, sondern relativ wertlos. Eine Replikation mit einer identischen Stichprobe überprüft, ob das Modell stichproben - ideosynkratisch gefittet hat (das hattest du angesprochen) - eine Replikation in einer anderen Stichprobe oder veränderten Bedingungen testet die Generalisierbarkeit.
Sehe ich nicht so, da man bei der adäquaten Interpretation von Parametern immer unterstellt, dass die spezifizierten Modelle valide die Kausalstruktur abbilden. Den Begriff Generalisierbarkeit habe ich bewusst vermieden, da man im deutschen Sprachgebrauch auf die Idee kommen könnte, dass man die externe Validität durch Replikation überprüft. Tatsächlich dient Replikation in der von mir benutzten Bedeutung der Überprüfung der interne Validität, sprich der Gültigkeit der Kausalstruktur gegeben der (Sub-)population für die das Modell Gültigkeit besitzen soll (wohlgemerkt keine ideosynkratisch Testung). Diese ist eine Grundbedingung für die Bewertung von Model-Fit. Ohne die Annahme interner Validität wäre statistische Inferenz in jeglicher Form überflüssig und unnütz.
Holgonaut hat geschrieben:Ein misspezifiertes aber fittendes Modell wird auch in einer neuen Stichprobe wieder fitten. Wenn du z.B. ein Modell genau so wie du es kritisiert hast, "verschönt" durch absurde Parameter und es so noch misspezifierter wird, und du testest es in einer neuen Stichprobe, wird es wieder fitten.
Falsch (siehe auch oben). Für weitere Erörterung wäre es notwendig, dass du dir die einschlägige Literatur zu Gemüte führst, in der divergente Interpretationen aufgrund konventioneller Fitindizes auch ganz praktisch anhand von Beispielen dokumentiert werden.
Holgonaut hat geschrieben:Was wenn das korrekte Modell nicht sparsam ist? Dann ist das "weniger komplexe Modell" misspezifiziert. "Zu gut fittend" gibt es m.E. nicht. Allerdings sehe ich es (auch) so, dass *saturierte* Modelle (meinst du das mit komplex?) ein kleineres Falsifikationspotential haben - d.h. ein saturiertes Modell mit gutem fit hat eine geringere Überzeugungskraft, weil die eben Gefahr besteht, dass man mit unsinnigen Parametern, den (sonst sichtbaren) misfit kompensiert. Nur hat das m.E. Implikationen für das Design (--> Modelle so spezifizieren, dass sie Restriktionen beinhalten) - nicht für die post-hoc-Anwendung von BIC & Co. Ich bin z.B. ein großer Fan von Instrumentalvariablen, weil diese genau diese essentiellen Restriktionen bieten.
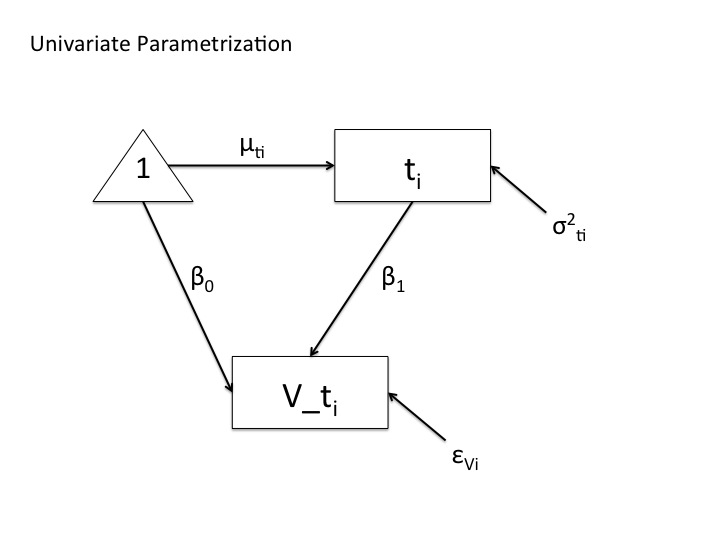
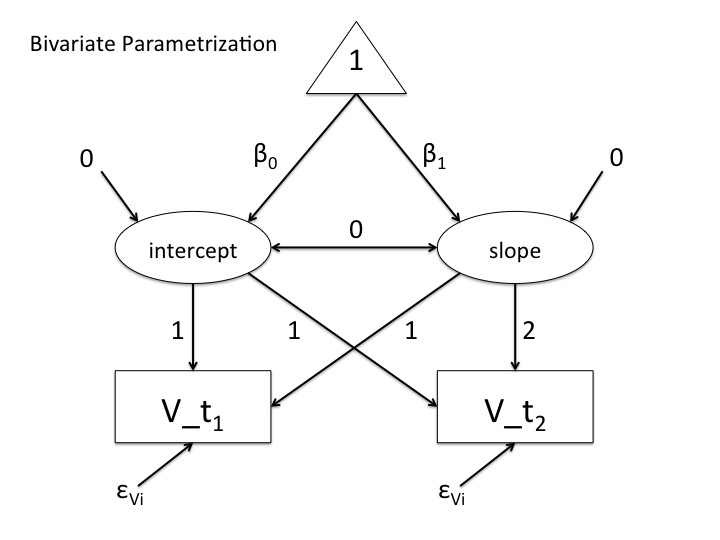
Hab mal 2 Pfaddiagramme fertig gemacht und angehangen – verschiedene Modelle, die identische Parameterschätzungen erzeugen, nur mit dem Unterschied, dass das univariate Modell gesättigt ist, das andere nicht. Wenn man sich das multivariate Modell anschaut stellt man anhand von chisquare basierter Fitindizes fest, dass es die Datenstruktur nicht adäquat replizieren kann, das univariate hingegegen perfekt. Beide Modell haben gleiche logLikelihood und gleiches AIC. BIC ist bei großen Stichproben auch asymptotisch gleich. Fittet das Modell die Daten jetzt erschöpfend oder nicht?
Holgonaut hat geschrieben:Interessante Diskussion by the way
Dito
Gruß, R