Hallo zusammen!
Ich habe die Ergebnisse eines GLM bekommen und muss diese nun schriftlich präsentieren. Leider hatte ich bisher in Sachen Statistik nur mit einfacheren Tests und bin daher froh, wenn ich einigermaßen verstehe, was das Modell berechnet. Nun weiß ich aber nicht so recht, wie die Ergebnisse präsentieren soll, bzw. Welche Werte alle zwingen aufgeführt werden müssen.
Kann mir da jemand weiterhelfen?
Ich hatte schon versucht Arikel zu finden, die damit arbeiten um die Methode zu übernehmen, finde dabei aber nur solche, die über das GLM diskutieren, aber nicht selbst damit arbeiten.
Hier mal ein Satz ergebnisse, wie er aus R kommt. Vielen Dank schon mal im Vorraus.
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: binomial ( logit )
Formula: gone ~ owl + attack + (1 | roost) + (1 | IDbat)
Data: owlattack
AIC BIC logLik deviance df.resid
805.9 829.6 -397.9 795.9 849
Scaled residuals:
Min 1Q Median 3Q Max
-1.1021 -0.4852 -0.3743 -0.2596 4.6706
Random effects:
Groups Name Variance Std.Dev.
IDbat (Intercept) 0.6867 0.8287
roost (Intercept) 0.8864 0.9415
Number of obs: 854, groups: IDbat, 46; roost, 8
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.6153 0.5227 -1.177 0.23916
owl -0.1955 0.2191 -0.892 0.37239
attack 0.9148 0.3494 2.618 0.00884 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) owl
owl -0.078
attack 0.043 -0.419
Ergebnisse aus GLM präsentieren
Thema bewerten: 




 • 2 Beiträge
• Seite 1 von 1
• 2 Beiträge
• Seite 1 von 1
- Harkon
- Grünschnabel

- Beiträge: 2
- Registriert: Mo 14. Nov 2016, 17:37
- Danke gegeben: 1
- Danke bekommen: 0 mal in 0 Post
Re: Ergebnisse aus GLM präsentieren
Ist zwar schon älter, aber falls es trotzdem noch jemanden interessiert, ich habe mich bei meiner Abschlussarbeit entschieden, neben den Model-Parametern (Steigung, Standardabweichung, Freiheitsgrade nach Satterthwaite Approximation, t-Wert, Wahrscheinlichkeit für diesen t-Wert) als wichtigstes Kriterium das adjustierte marginale und konditionale 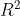 nach der Methode von Nakagawa et al. anzugeben. Diesen Wert und andere hilfreiche Maße für die Qualität eines lmm erhält man mit R einfach durch die "piecewiseSEM" library mit dem Befehl "sem.model.fits(MODELNAME, aicc=T)"
nach der Methode von Nakagawa et al. anzugeben. Diesen Wert und andere hilfreiche Maße für die Qualität eines lmm erhält man mit R einfach durch die "piecewiseSEM" library mit dem Befehl "sem.model.fits(MODELNAME, aicc=T)"
Nakagawa, Shinichi, Holger Schielzeth, and Robert B. O’Hara (2013). “A general and simple method for obtaining R2 from generalized linear mixed-effects models”. In: Methods in Ecology and Evolution 4.2, pp. 133–142. ISSN: 2041210X.
Nakagawa, Shinichi, Holger Schielzeth, and Robert B. O’Hara (2013). “A general and simple method for obtaining R2 from generalized linear mixed-effects models”. In: Methods in Ecology and Evolution 4.2, pp. 133–142. ISSN: 2041210X.
- StefanM
- Grünschnabel

- Beiträge: 2
- Registriert: Mo 12. Feb 2018, 11:59
- Danke gegeben: 0
- Danke bekommen: 0 mal in 0 Post
Thema bewerten: 




 • 2 Beiträge
• Seite 1 von 1
• 2 Beiträge
• Seite 1 von 1
Wer ist online?
Mitglieder in diesem Forum: 0 Mitglieder und 8 Gäste


